Abstract
특허 출원이 다루는 화학 및 생물학적 공간을 탐색하는 것은 초기 단계의 의약 화학 활동에서 매우 중요하다. 특허 분석은 화합물 선행 기술에 대한 이해, 신규성 확인, 생물학적 분석의 검증 및 화학적 탐색을 위한 새로운 출발점의 식별을 제공할 수 있다. 전문 큐레이터가 직접 특허에서 화학적 및 생물학적 entity를 추출하는 데는 상당한 시간과 리소스가 소요된다. 텍스트 마이닝 방법은 이 프로세스를 용이하게 하는 데 도움이 될 수 있다. 이러한 방법의 성능을 검증하려면 수동으로 annotation을 추가한 특허 자료가 필수적이다.
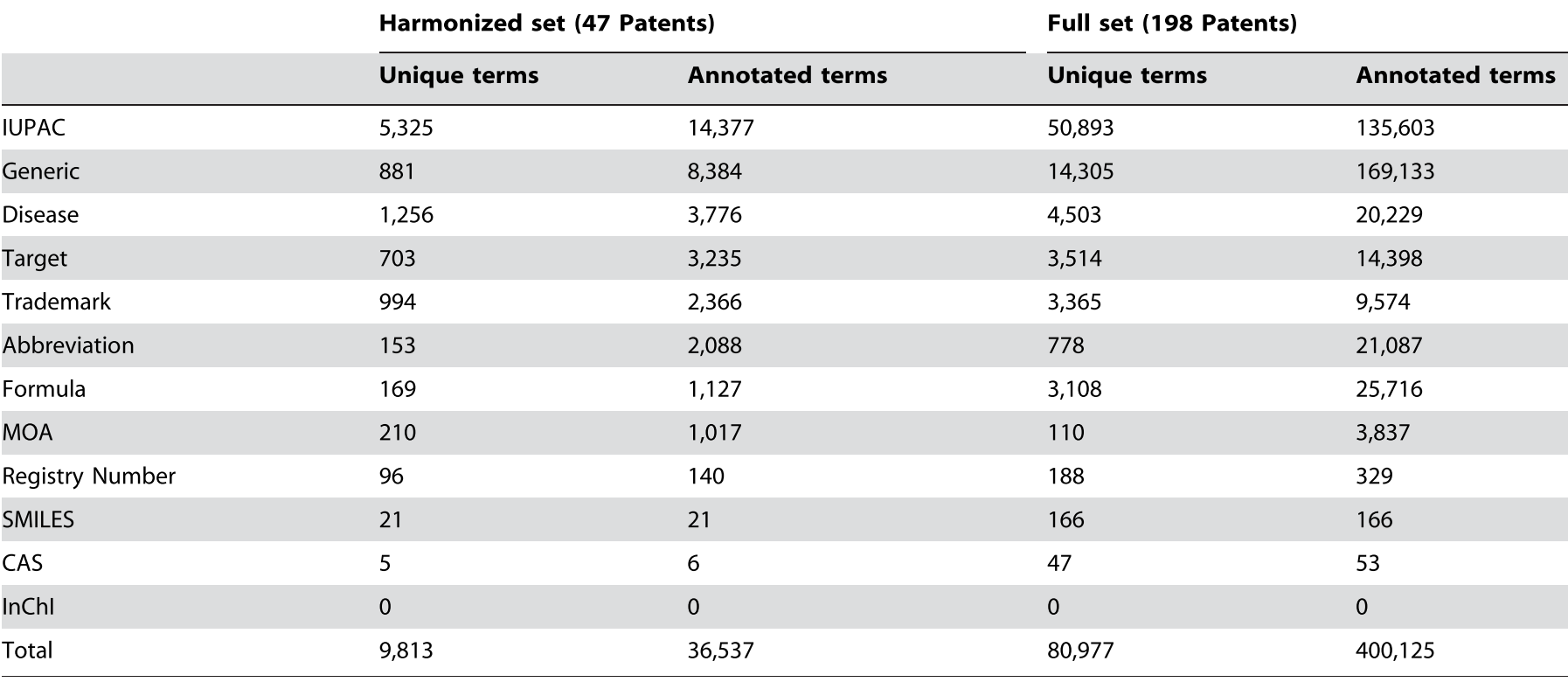
우리는 annotation guideline을 개발하고 세계 지적 재산권 기구, 미국 특허청, 유럽 특허청에서 200개의 전체 특허를 선택했다. 특허는 사전에 자동으로 주석이 달렸고 각각 2~10명의 annotator로 구성된 4개의 그룹에서 사용할 수 있게 했다. Annotator는 chemical을 각각 다른 subclasses, diseases, targets, modes of action으로 구분했다. 광학 문자 인식(OCR) 오류로 인한 맞춤법 오류 및 잘못된 줄 바꿈도 주석 처리되었다. 47개 특허의 하위 집합에 적어도 3개의 annotator 그룹이 주석을 달았으며, 이 그룹에서 harmonized annotations과 inter-annotator agreement scores가 도출되었다. 한 그룹은 전체 세트에 주석을 달았다. patent corpus에는 전체 집합에 대한 400,125개의 annotation과 조화된 집합에 대한 36,537개의 주석이 포함된다. 모든 특허 및 주석이 달린 엔티티는 www.biosemantics.org에서 공개적으로 사용할 수 있습니다.
1. Introduction
신약 개발 단계에서 화학 특허 탐색은 사전 기술, 신규성 확인, 생물학적 분석의 검증, 화학적 탐색의 새 시작점 확인을 위해 중요하다. 특허 내의 chemical and biological entity 추출은 복잡하여 사람이 직접 하거나 text mining의 도움을 받기도 한다. The European Patent Office(EPO), United States Patent and Trademark Office(USPTO), World Intellectual Property Organization(WIPO)는 온라인으로 접근이 가능하며 무료로 XML, HTML, PDF 형태로 파일을 받을 수 있다. PDF의 경우 OCR을 통해 추출할 수 있으며 사실 HTML과 XML도 특허 사무소에서 OCR을 이용해 추출한 것이다.
그러나 text mining은 다수의 많은 화학물 이름, 모호한 용어, 복잡한 구문구조, OCR 오류로 어려움이 많다.
Named entity recognition 성능을 검증하기 위해 manually annotated patent corpus가 필요하다. 그러나 이러한 annotated text 생성에는 많은 노력과 비용이 들기 때문에 대부분 화학이 아닌 유전자와 단백질 관련 corpus 생성에만 노력이 집중되었다.
Corpus 개발 history
- 2003 GENIA:여러 종류의 화학 물질로 구성 (Kim et al.)
- 2004 BioIE: 화학 물질 및 단백질에 대한 주석을 포함 (Kulick et al.)
- 2008: 화학 화합물로 주석이 달린 과학 초록의 작은 corpus (Kola'rik et al.)
- 최근 CHEMDNER: 다양한 종류의 화학 물질로 annotation, BioCreative 챌린지의 일부로 제공
이 모든 corpus는 Medline의 scientific abstract로 구성된다.
2009년 EPO와 ChEBI(Chemical Entities of Biological Interest) 간의 공동 프로젝트에서 화학 물질을 포함하고 annotation이 가능한 화학특허의 경우 ChEBI 화합물에 대한 매핑을 포함하는 화학 특허 corpus가 개발되었다. 이후 지속적으로 발전시켜 2012년 큰 규모의 일반 화학물의 NER을 포함하는 특허 corpus를 Kiss et al.에 의해 개발됐다.
이전까지 특허의 복잡성과 길이 때문에 gold standard patent corpus는 체계적으로 다루어지지 않았다. 이전 시도에서는 제한된 수의 화학 물질에만 주석이 달렸고 subclass는 정의조차 되지 않았다. disease나 mode of action과 같은 다른 생물학적 entity는 포함되지 않았으며 철자나 OCR로 인한 오류는 고려되지 않았다. 이전 corpus 연구들은 inter-annotator agreement에 대한정보를 제공하지 않으나 이는 text mining 프로그램의 성능을 평가하고 비교하는데 유용하다.
해당 논문에서는 text mining 성능 bechmark를 위한 200개의 전체 patent의 gold standard annotated corpus를 제공한다.
2. Methods and Materials
- Corpus development strategy
1. annotation guideline을 개발, 200개의 다양한 특허 set를 선택
2. 자동으로 pore-annotation 수행, 독립적인 네 개의 annotator 그룹에게 제공
3. annotation guideline 개발을 위해 두 개의 특허를 사용. 나머지 특허는 47개의 특허 subset을 최소 3개의 그룹에 의해 annotation이 추가되는 방식으로 여러 annotator group에 배포
4. harmonized set의 inter-annotator agreement score를 통해 group간 비교. 하나의 group이 하나의 set를 annotate
- Patent corpus selection
GVK BIO target class databse를 patent corpus selection을 시작점으로 사용했다. EPO, USPTO, WIPO 모두 해당 DB를 통해 접근 가능하다. 해당 DB는 문서 간 관계, assay, 화학 구조, assignees, protrin targets를 포함하며, protein families를 기반으로 분류된다. 10 ~ 200 개 사이의 예시 화합물을 포함하는 모든 영어 특허와 명명된 기본 대상이 GVK BIO DB에서 선택됐다. 모든 화합물의 문자량이 1000 미만인 특허만 선별했으며, 기간에 대한 제한은 지정하지 않았다. 총 28695개의 특허가 위의 기준을 충족했다. 각 회사마다 특허 작성 방식이 다르기 때문에 만약 동일한 assignee가 primary target에 대해 다수의 특허를 작성했다면 임의로 하나만 취하고 나머지는 무시했다. 위 기준을 토대로 11개 target classes에 대해 8016개의 특허가 남았다. 유명한 특허는 포함시키기 위해 50개의 약물 특허를 추가했다. 최소 10개의 특허가 있는 각각의 group에서 특허를 임의 추출했다. 다양성은 Table 1. 에서 확인할 수 있으며, final set 은 121개의 USPTO, 66개의 WIPO, 13개의 EPO를 포함하고 11500 pages와 4200만개의 단어를 포함한다.
특허들은 XML 형태로 다운 받았으며, 연속되는 줄바꿈은 하나의 줄바꿈으로 대체 되었다. (아마, \n\n... → \n) 또한 모든 Image들은 제거되었다.
- Annotated entities
특허 내의 모든 compounds, diseases, protein targets, modes of actions(MOA)는 전부 annotated 했다.
Compounds, 생성 방식에 따라 systematic identifiers와 non-systematic identifier로 annotated.
*Systematic identifiers
> IUPAC, SMILES, InChI,
*Non-systematic identifiers
> tradesmarks (Aspirin, Mesupron), abbreviations (DCM, TBTU), CAS number, formulas(MgSO4), generic names (cycloalkylamines)
Diseases (diabetes), Protein target (trypsin), MOAs (antagonist)
OCR 오류 (철자, line break)도 annotate 했다.
- Annotated guidelines
이전 연구의 guideline을 기반으로 진행되었으며, 임의로 뽑은 두 patents (US5023269, US4659716)를 기반으로 annotator를 학습 및 guideline을 구축했다.
1. Entity가 중첩되거나 다른 개체와 겹치는 경우 더 구체적인 entity에 주석을 답니다. 예를 들어 ''5-HT1D''는‘‘5-HT1D Serotonin Receptors’’의 대상 주석 내에 포함된 경우 대상으로 주석을 추가해서는 안 됩니다.
2. ‘‘water, ‘‘ammonia’’, and ‘‘ethanol’’과 같은 간단한 IUPAC 이름에 주석을 답니다.
3. 주석 내에 접두사를 포함해야 합니다(예: ''1,4-butanediol''의 ''1,4-'').
4. 'NaOH' 및 '(NH4)2SO4''와 같은 간단한 formulas는 formulas으로 주석을 달아야 합니다.
5. ‘‘acetate’’, ‘‘oxalate’’, ‘‘propionate’’와 같은 counterions은 IUPAC 이름으로 주석을 달아야 합니다.
6. ‘‘4-halo-phenol’’ or ‘‘xylene’’과 같은 generic structures는 generic name으로 주석을 달아야 합니다.
7. Polymers, e.g., ‘‘Polystyrene’’는 일반 이름으로 주석을 달아야 합니다.
8. ‘‘Sildenafil’’ 같은 간단한 이름은 IUPAC 이름으로 주석을 달아야 합니다.
9. ‘‘include inorganic acids such as hydrochloric, hydrobromic’’의 ‘‘hydrochloric’’ 및 ‘‘hydrobromic’’과 같은 열거는 IUPAC 이름으로 주석을 달아야 합니다.
10. ''N'', ''O'', ''C''와 같은 요소는 주석 처리되어서는 안 됩니다.
11. 철자가 틀린 용어는 spelling mistakes로 주석을 달아야 합니다(예: ''hydrobroml:c'').
12. 가짜 줄 바꿈으로 인해 여러 줄에 걸쳐 있는 주석은 하나의 용어로 주석을 달고 가짜 줄 바꿈으로 태그를 지정해야 합니다.
13. 추가 공백은 철자 오류로 주석을 달아야 합니다(예: "hydro bromic'').
14. OCR 오류 이외의 사유로 분할된 용어는 주석을 달지 마십시오.
15. 쉼표, 전하 기호 또는 대괄호와 같은 모든 기호는 주석에 포함되어야 합니다(예: ''n1c[nH]cc1'').
- Annotated process
Leadmine 이라는 NextMove社의 S/W를 이용하여 pre-annotation을 진행했다. LeadMine은 chemicals, protein targets, genes, species, company names 그리고 오자도 잡아 낼 수 있다. LeadMine으로 가능한 annotation만 처리했으며, SMILES, InChI, Diseases, MOAs는 LeadMine으로 처리가 불가능하다. Annotation은 Brat rapid annotation tool(v1.3)을 이용하였다. 성능 때문에 특허를 Brat에 표시하기 위해 50개 단락이 있는 페이지로 분할했다.
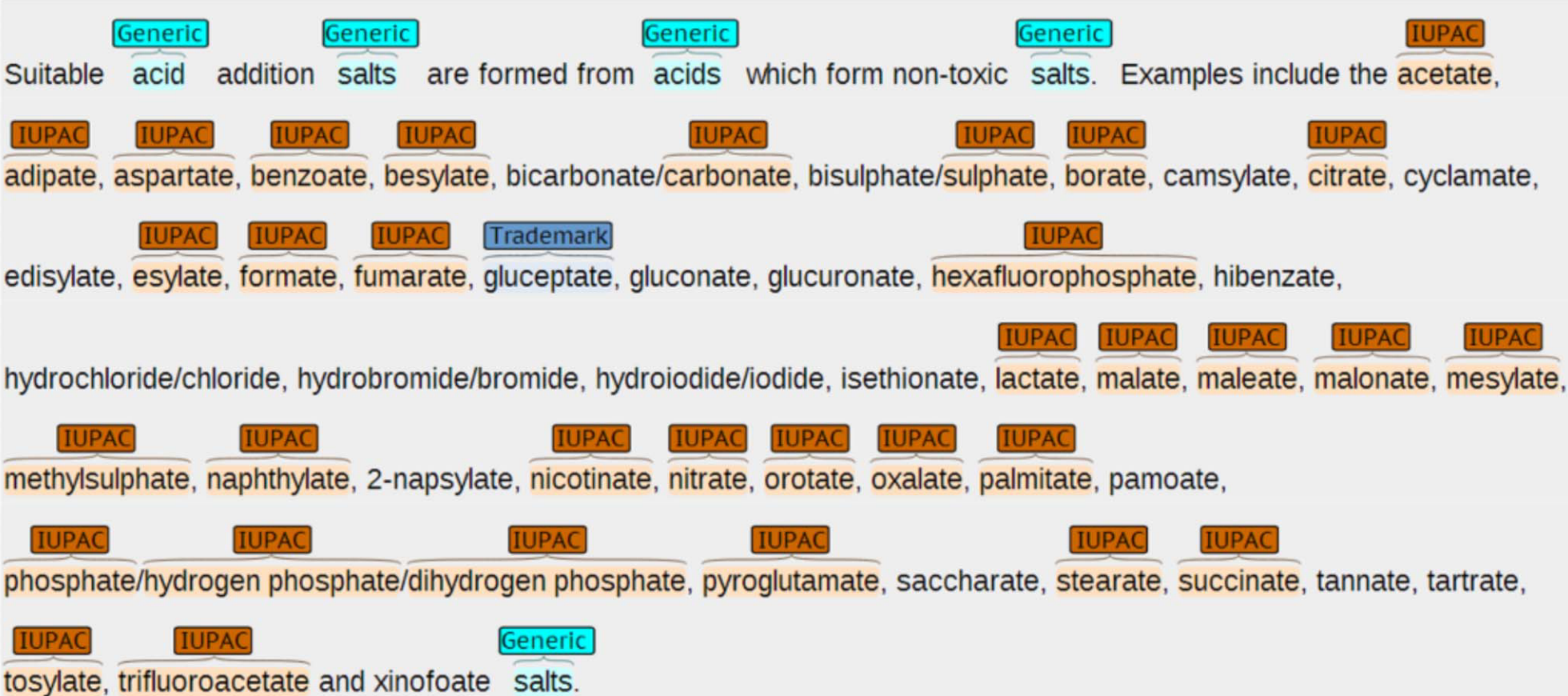
이렇게 전처리된 특허들은 네 그룹에게 전달 됐다. (AstraZeneca, Fraunhofer, GVK BIO, and NextMove) GVK BIO가 10명인 것을 제외하고 각각 2명의 annotator로 group은 구성됐다. Fraunhofer는 pre-annotation을 무시했다. 각 특허는 group 내의 한 명의 annotator에게 전달되어 작업을 수행했다.
- Resolving misannotation of ambiguous terms
모든 그룹의 주석 완료 후 annotation group은 corpus 내에서 모호한 용어의 수를 줄이기 위해 결과를 검토했다. 다른 그룹이 말뭉치 전체에서 다른 entity 유형으로 용어에 주석을 추가한 경우 해당 용어는 모호한 것으로 정의된다. 모호하게 주석이 달린 용어의 목록이 컴파일되었고 주석가는 각 모호한 용어에 할당된 서로 다른 엔티티 유형을 기반으로만 목록을 검토하도록 요청했다(즉, 용어의 컨텍스트가 제공되지 않음). 주석가는 각 용어를 세 개 중 하나로 분류해야 했다.
1- 해당 용어에 할당된 entity 유형이 없다. 용어의 모든 주석이 corpus에서 제거되었다. ("nitrogen"은 원소이므로 IUPAC도 Generic도 아니다)
2- 하나의 엔티티 유형이 적용. corpus 내에서 발생하는 모든 용어가 이 엔터티 유형에 할당된다. ("DMF"의 경우, Trademark로 43번 Abbreviation으로 289번 annotated 되었지만, 문맥에 상관없이 DMF는 abbreviation이므로 전부 이로 할당했다)
3- 둘 이상의 엔티티 유형이 적용된다. 적용할 수 없는 항목 유형이 있는 주석만 corpus 전체에서 제거되었다. ("5-ht"라는 용어는 Abbreviation으로 17번, Generic으로 25번, Target으로 23번 주석을 달렸다. 텍스트의 문맥에 따라 용어는 Target 또는 Abbreviation일 수 있지만 Generic은 아니기에 Generic은 corpus에서 제거되었다.
- Harmonization
Gold standard corpus를 위해 47개의 특허를 세 group에게 annotation을 시켜 하나의 harmonized set으로 만들었다. Centroid algorithm(Lewin et al.)이 사용됐다.
해당 알고리즘은 annotation을 character level로 tokenize한 후 인접한 annotator간 annotation의 중복된 pair의 character 수를 샌다. 개별 character가 아닌 annotation 내부 character pair에 voting을 통해 두 용어가 서로 직접 인접하여 주석이 달린 상황에서 경계(주석이 있는 엔터티 유형의 시작 및 끝 위치)가 고려된다. Harmonized annotation은 지정된 threshold보다 크거나 같은 투표를 갖는 character pair로 구성된다. 이 작업에서 우리는 2를 투표 임계값으로 사용했다. 즉, 적어도 두 명의 annotator가 annotation에 동의해야 했다.
- Inter-annotator agreement
F-score를 이용해 Inter-annotator agreement를 판단하였으며 gold standard set과 비교하였다. 만약 entity type과 start, end 지점이 같으면 gold standard와 동일한 것으로 취급했다. Gold에 없는데 표기했으면 false negative, 정확하게 일치하지 않으면 false positive로 취급했다.
3. Results
- Patent distribution among group
각각 NextMove社 27개, Fraunhofer社 36개, AstraZeneca社 49개, GVK BIO社 198개 의 특허를 annotation
그 중 47개의 특허가 최소 3개의 group에서 annotation 했다. (4개 group 모두 annotation 한 특허는 총 3개였다)
- Initial harmonized set
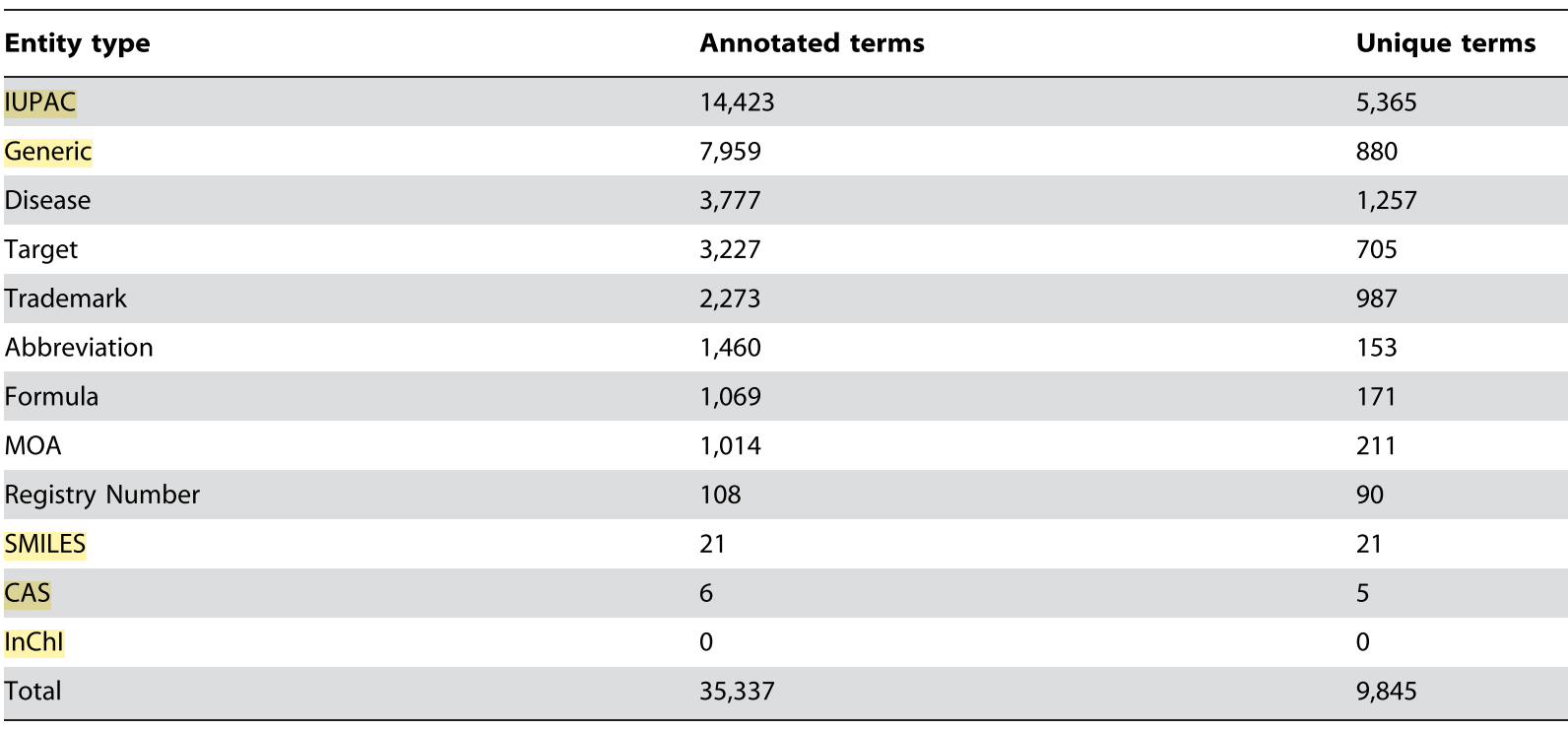
47개의 특허를 기반으로 총 35337개의 주석이 존재. 대부분이 IUPAC과 Generic이며, SMILES, CAS#, InChI는 거의 등장하지 않았다.
- Inter-annotator agreement

- Disambiguagtion
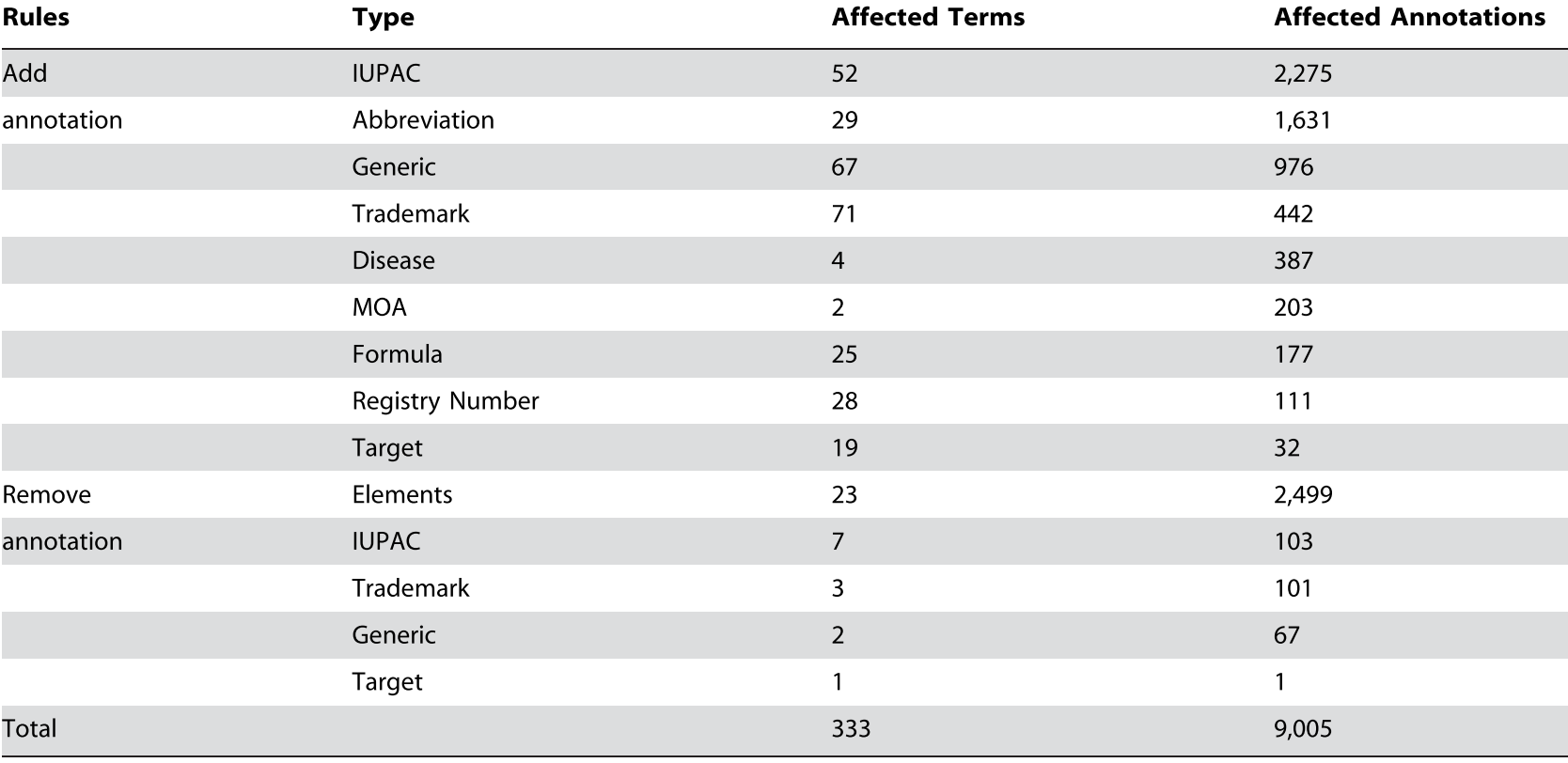
47,044개의 주석에 해당하는 2,135개의 고유한 모호한 용어 세트가 위에서 설명한 대로 명확성을 위해 주석자에게 제공되었다. 주석 작성자는 333개의 고유한 모호한 용어에 대한 결정을 내릴 수 있었고, 이는 9,005개의 주석에 영향을 미쳤다. 대부분은 IUPAC 이였으며, 23개의 elements도 총 2499번 다른 타입으로 주석 annotation 되었다.
- Inter-annotator agreement after disambiguation
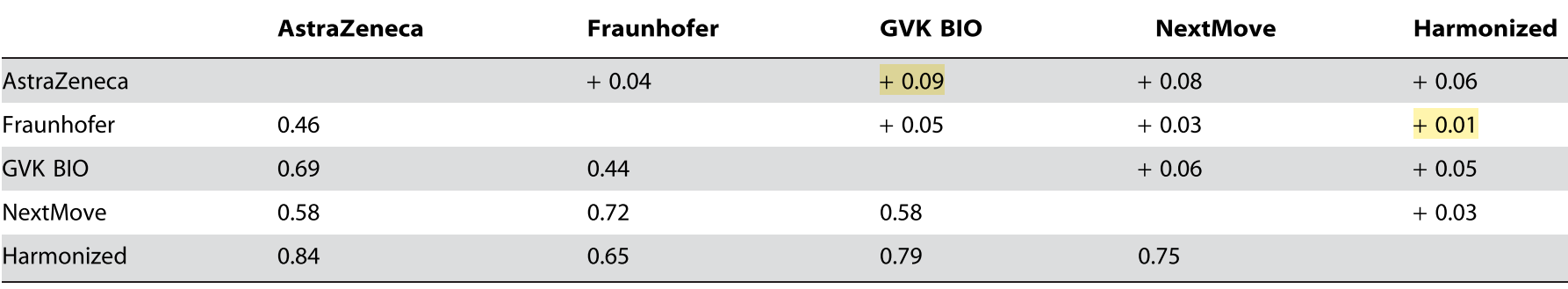
Disambiguation을 통해 0.01에서 0.09 points IAA 향상이 있었다.
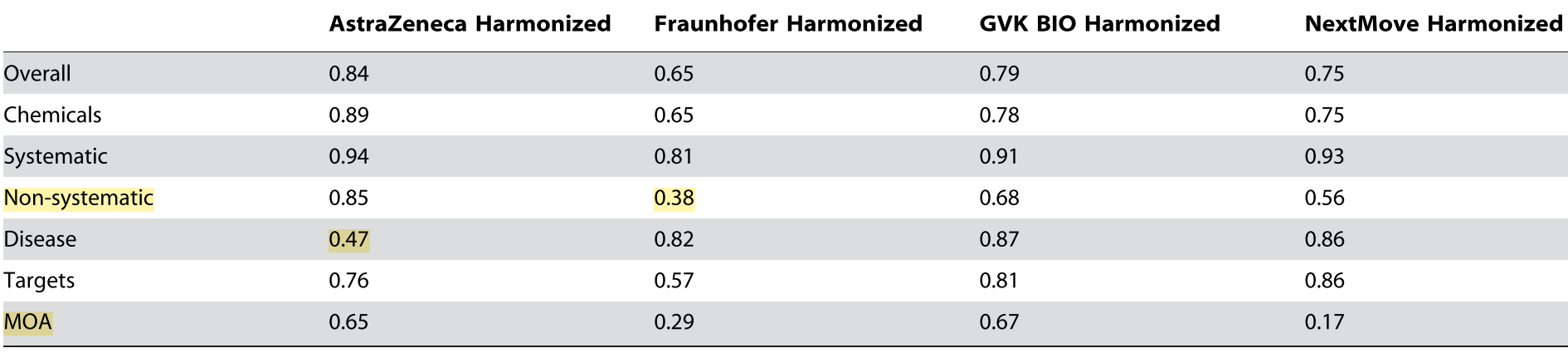
Annotation의 주요 어려움은 non-systematic identifier 및 MOA에 대해 발생했으며 targets, diseases 및 systematic identifier의 식별은 더 높은 agreement로 이루어졌다.
- The gold standard patent corpus